评估NGS库准备的输入RNA的质量
RNA-sequencing(RNA-Seq)已成为转录组分析的越来越流行的技术[1]。高质量RNA-seq库的制备对于测序数据的可靠性至关重要,其中输入RNA的完整性和纯度起着至关重要的作用。因此,在进行图书馆准备之前,对输入RNA进行质量控制是一种好习惯。在这里,我们提供了一些最佳建议,以帮助保存和验证输入RNA的质量,以获得更强大的RNA-Seq库。
关于RNA提取
降解的RNA可以偏向基因表达的测量,提供不均匀的基因覆盖范围,并防止分化替代剪接的转录本[2]。因此,建议尽可能将高融合性RNA用作输入。处理RNA样品时:
- 收集后立即从样品中提取RNA,尽可能减少细胞RNase活性。如果需要临时样品存储,请在稳定解决方案中保存样品,例如DNA/RNA盾牌或Trizol®,作为这些产物的裂解特性有效地取代了细胞RNass。德赢vwin体育官方网址
- 在整个工作流程期间,请使用无菌过滤器尖端,以最大程度地减少RNase并进行跨样本污染。
- 在使用时将纯化的RNA保持在冰上,并将其存储在-80°C中,以减少冻融周期。
- 用DNase I处理RNA样品,以消除污染的DNA。DNA的存在可能会引入生物信息学处理和结果库的数据分析。
评估可靠NGS库准备的输入RNA的质量
在输入RNA上执行质量控制(QC):
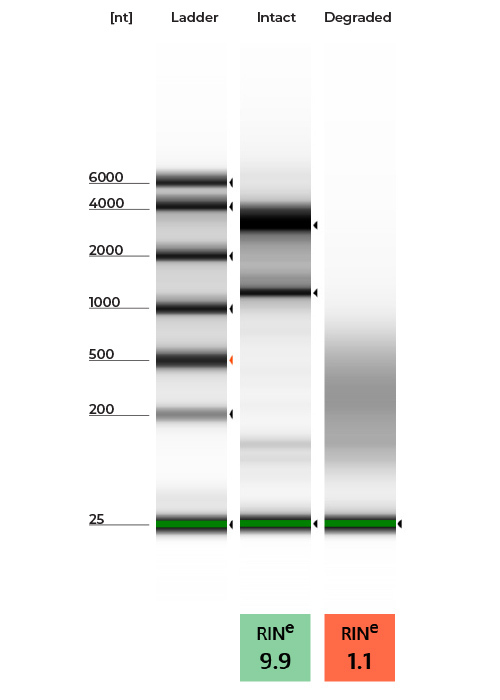
- 通过获得片段大小和RNA完整性数(RIN)来表征RNA的完整性。高质量的RNA将具有两个突出的,缓慢的迁移带,代表RRNA片段(例如HeLa细胞的18s和28s)。涂抹和较小片段的突出表明RNA降解。
- 通过首选方法(例如Agilent的挂毯)确定片段大小和RIN(图1中显示的示例数据)。
- 另外,使用传统的凝胶电泳也可以以相似的方式提供尺寸分布信息(图像类似于图1A)。
- 使用UV-VIS分光光度计(例如Nanodrop™)评估输入RNA的纯度,以确保RNA不受污染的苯酚和混合盐的污染。对于纯RNA,理想的A260/A280比率约为2.0,理想的A260/A230左右为2.0-2.2。
一种

b
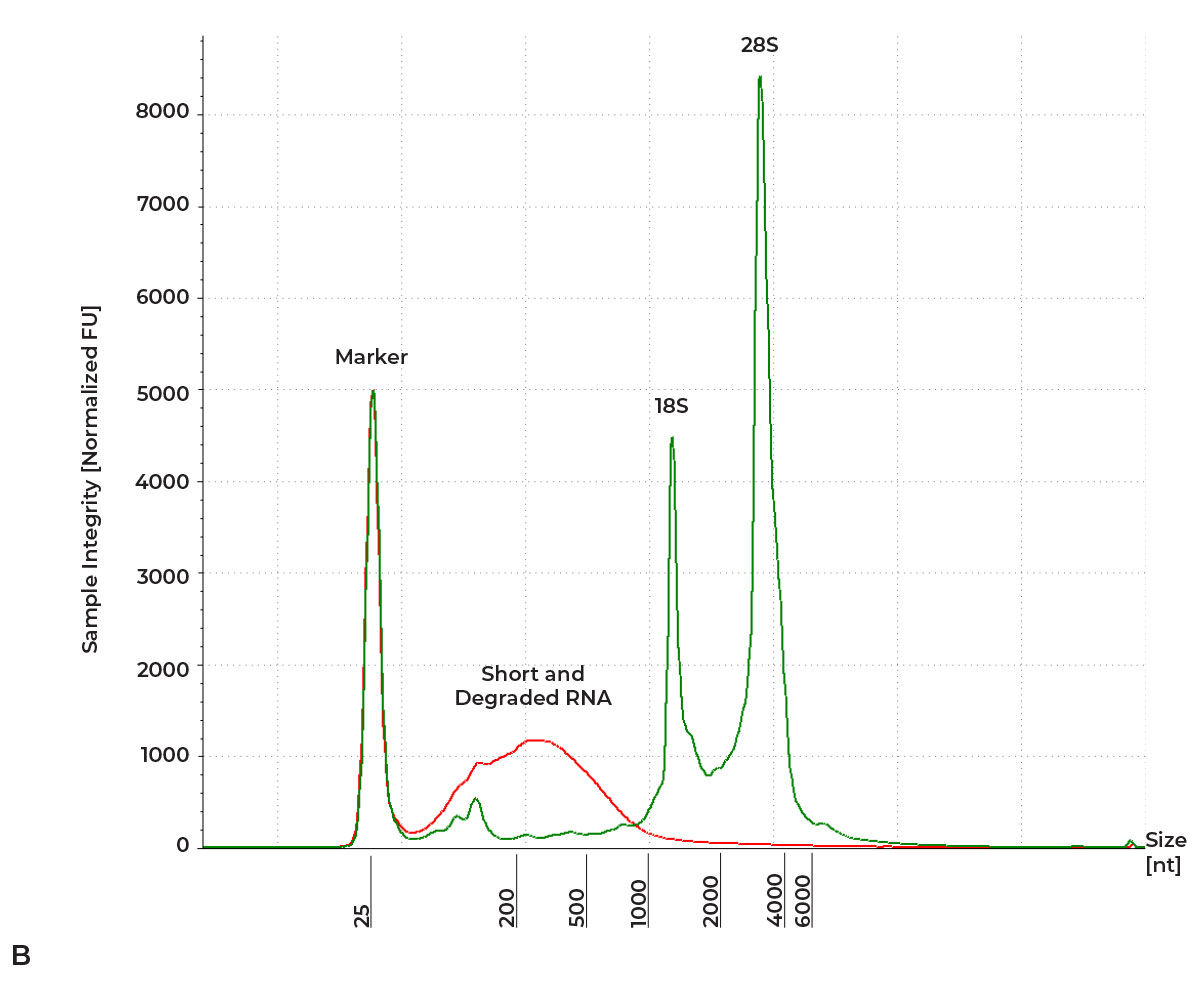

图1:A)高质量的RNA与降解的RNA凝胶图像。b)高质量的RNA与降解的RNA电池图。高质量和完整的RNA由两个缓慢移动带(绿色)表示表示。相比之下,降解的RNA由短而快速移动的片段(红色)的积累表示。RNA在带有4150挂号系统的RNA屏幕板上进行了特征。
遵循这些技巧和技巧可以帮助您准备适合RNA-seq的RNA样品并产生可靠的测序数据。
了解有关Zymo的RNA NGS图书馆准备产品的更多信息
学到更多参考
- Hong,M。;Tao,s。;张,L。;Diao,L。T。;黄,x。Huang,s。;Xie,S.J。;Xiao,Z。d。;Zhang,H。RNA测序:癌症研究中的新技术和应用。J Hematol Oncol 2020,13(1),166。DOI:10.1186/S13045-020-01005-X。
- K. R. Kukurba;Montgomery,S。B. RNA测序和分析。冷弹亨布尔布原始2015,2015(11),951-969。doi:10.1101/pdb。TOP084970。
- W. W. Wilfinger;Mackey,K。;Chomczynski,P。pH和离子强度对核酸纯度分光光度法评估的影响。Biotechniques 1997,22(3),474-476,478-481。doi:10.2144/97223ST01。
- unger,c。;Lokmer,N。;雷曼,D。Axmann,I。M. RNA样品中苯酚污染的检测及其对QRT-PCR结果的影响。肛门生物化学2019,571,49-52。doi:10.1016/j.ab.2019.02.002。
- Shen,C.-H。核酸的检测和分析。在诊断分子生物学中,Chang-Hui,S。Ed。;学术出版社,2019年;第167-185页。


