裂解偏见危机
打破错误
从微生物细胞中解放DNA可能非常困难。例如,由于各种原因(细胞壁厚度,复杂的脂质,多糖等),某些细菌细胞类型对化学裂解是难治性的。此外,其他微生物,例如机会性人类病原体,加密型新羊角,众所周知,很难裂解,甚至显示出对化学裂解技术响应次级细胞壁的能力[1]。此外,复杂的微生物群落很可能包含类似的很难散布的物种,并且如果这些顽固的微生物没有与易于散热的物种相同的效率,则不可能具有准确的特征表征。
过分代表的震惊
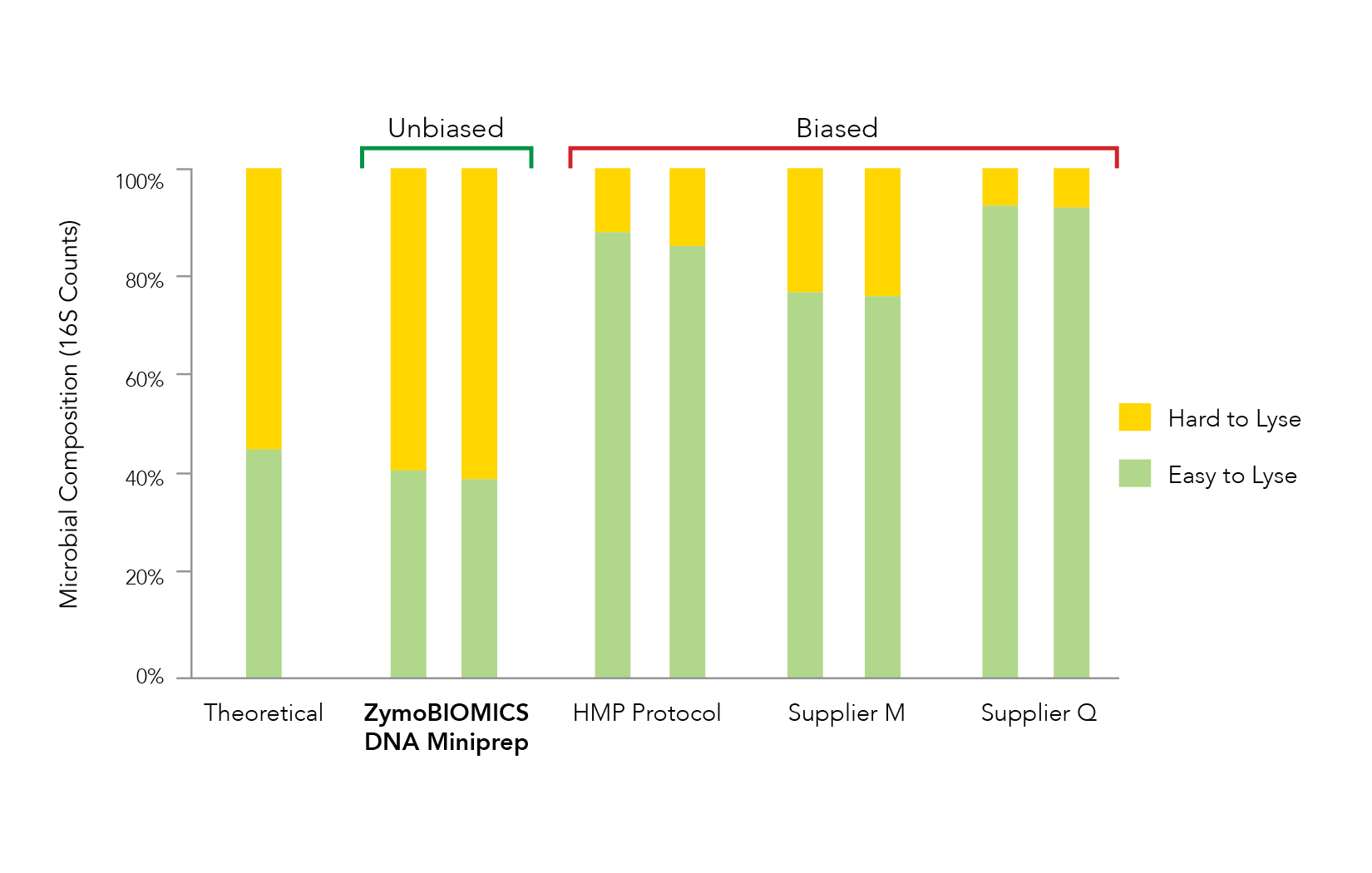
准确的微生物组分析需要从微生物群落中的所有不同物种中解放DNA。不幸的是,通常观察到核酸提取过程中无效的裂解,从而导致微生物谱偏置(图1)。和更多的科学家意识到偏见对微生物学构成的威胁,正在研究和优化许多不同的细胞裂解方法。这些包括机械,化学,热和酶促技术。尽管所有方法都有缺点,但所有方法的优化都需要针对定义的微生物社区进行严格的基准测试标准确保准确表示易于散热和易于散热的微生物。
已经表明,利用化学或热裂解的过程通常会导致易于散热生物(例如革兰氏阴性细菌)的过度占代表性,这是由于DNA从坚硬的生物体中释放而产生的(例如,革兰氏阳性细菌和酵母)[2-3]。此外,酶裂解本质上是固有的,因此很容易受到偏见的影响,尤其是从高度多样化的样本类型(例如土壤)中。
Deus Ex机械
机械裂解方法(例如超声处理,液氮/砂浆和杵,法式压力和珠子跳动)被认为是微生物裂解的最佳方法,因为它们的随机性质,珠子的跳动通常称为金标准。但是,如果未优化,机械裂解方法可能会遭受诸如低产量,过度核酸剪切,由于不完全裂解而导致的非均匀裂解等问题,分别对易于渗透的微生物的效率低下。如果未验证珠子跳动协议以克服这些问题,则可能会偏向生成的微生物剖面。不幸的是,尽管有可能提高产量,但简单地结合一系列细胞裂解机制并不一定会降低偏差。
致力于准确性
为了帮助所有微生物组研究人员产生最准确的微生物分析数据,Zymo Research评估了几种珠子跳动程序,并确定了产生无偏核酸提取物的最佳参数。使用Zymobiomics微生物社区标准还有唯一经过验证的无偏见DNA提取试剂盒,Zymobiomics DNA微型培养箱,使这些方法可以基准测试。Zymo Research热衷于确保Microbiomics领域具有创建准确和可重现数据的最佳研究工具。下面我们提供了使用Zymobiomics Microbial社区标准进行了广泛测试的Beat打击协议,并使用下一代测序对无偏见进行了验证。
随着人类健康,农业和环境安全的影响,微生物学研究生成准确和可重复的数据至关重要。由于偏置裂解技术而引起的LAB间可重复性差,困扰了微生物学领域,但是,我们正处于科学家寻求基准和标准化方法的转折点。通过利用经过验证的裂解参数,研究人员可以对其微生物分析数据的准确性更具信心。
使用Zymobiomics DNA Miniprep套件验证了珠子跳动方案:
MP FastPREP-24珠子跳动协议:
(带有2毫升bashingbead管)
最多20管。> 20管的重量可能会导致系统误差。
- 以最大速度1分钟
- 休息5分钟
- 重复周期5次,总计5分钟的珠子跳动
Biospec Mini-Beadbeater-96珠子跳动协议:
(带有2毫升bashingbead管)
- 5分钟在最大rpm处
- 休息5分钟
- 重复周期4次,总计20分钟的珠子跳动
Biospec Mini-Beadbeater-96珠子跳动协议:
(带有96个井立架)
- 最大rpm上的5分钟
- 休息5分钟
- 重复周期8次,总计40分钟的珠子跳动
Bertin旨在进化珠跳动方案:
(带有2毫升bashingbead管)
- 1分钟在9,000 rpm
- 休息2分钟
- 重复周期4次,总计4分钟的珠子跳动
Vortex Genie Bead Beating协议:
(带有水平(24)微管适配器)
最多18个试管。> 18管的重量将减慢机制并引入偏见。
- 40分钟的连续珠跳动(每个适配器最多18个试管)
参考:
1.FarkašV,Takeo K,MacekováD,Ohkusu M,Yoshida S,Sipiczki M.加密酮Neoformans中的次要细胞壁形成,是一种反对酸诱导的自动解的救援机制。FEMS酵母研究,2009,9(2):311-320
2. Sohrabi M,Nair RG,Samaranayake LP,Zhang L,Zulfiker AH,Ahmetagic A,Good D,Wei MQ。人口腔冲洗样品中细胞和细菌DNA提取物的产率和质量受细胞裂解方法的影响。微生物方法杂志2016,122:64-72
3. Yuan S,Cohen DB,Ravel J,Abdo Z,Forney LJ。评估从人类微生物组中提取和纯化DNA的方法。PLOS ONE 2012。


